


Интенсивное развитие методов РНК-секвенирования и увеличение количества публичных данных позволяют искать новые биомаркеры в больших когортах доноров или типов клеток для идентификации патологических состояний или клеточных линий. Одним из таких биомаркеров являются длинные некодирующие РНК (lncRNAs). Выявлено большое количество lncRNA: их число приблизилось к числу кодирующих РНК (17910 аннотированных lncRNA человека в последней версии референсного генома v32 GENCODE по сравнению с 19965 кодирующими генами). Все большее количество фактов указывает на возможность использования lncRNA как терапевтические мишени, а также как потенциальные тканеспецифические маркеры. Так, недавно были охарактеризованы специфические lncRNA, которые являются биомаркерами В-клеточного острого лимфобластного лейкоза и гипертрофической кардиомиопатии. Однако до сих пор не существует достаточно полного каталога lncRNA, связанных с определенной клеточной линией или патологией.
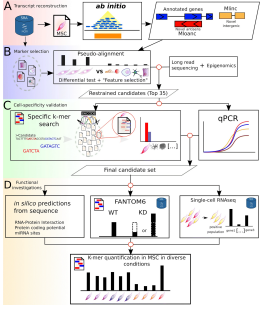
Ученые из университета Монпелье, Франция, разработали уникальный биоинформатический пайплайн для создания клеточноспецифического каталога неаннотированных lncRNA. Пайплайн выполняет ab initio идентификацию транскриптов, псевдовыравнивание и использует новые методики, такие как специфический подход k-mer для наивной количественной оценки экспрессии в многочисленных данных РНК-секвенирования. Пайплайн состоит из 4 этапов:
1) реконструкция транскриптов ab initio из данных РНК-секвенирования и характеристика новых транскриптов,
2) дифференциальный анализ с использованием псевдовыравнивания в сочетании с машинным обучением для извлечения наиболее специфичных для клеток кандидатов,
3) проверка экспрессии в тканях с поиском специфических k-меров в больших и разнообразных транскриптомных наборах данных,
4) углубленный анализ для предсказания функционального потенциала lncRNA с помощью подходов in silico предсказания.
Далее ученые проверили биомаркерный потенциал lncRNAs на мезенхимальных стволовых клетках, сложном типе мультипотентных стволовых клеток (МСК) различного тканевого происхождения. Полученные предсказания были валидированы с помощью количественной ПЦР в реальном времени и секвенирования Oxford Nanopore Technologies. Пайплайн смог выделить новые lncRNAs с высокой клеточной специфичностью. Особенно интересной находкой исследования является выделение трех новых lncRNA-маркеров МСК костного мозга и жировой ткани, которые прошли все этапы отбора и обладают интересными характеристиками: Mlinc.28428.2, Mlinc.128022.2 и Mlinc.89912.1.
Таким образом, подход может быть использован для выявления новых lncRNA в качестве клеточных маркеров, биомаркеров патологических состояний и предикторов течения заболеваний.